MATLAB实现基于神经网络MIV值分析的肿瘤基因信息提取 毕业论文+源码
题 目 基于神经网络MIV 值分析的肿瘤基因信息提取

摘 要:
本文主要运用统计学及数据挖掘相关知识,以结肠癌基因表达图谱为研究对象,综合运用 GB 指数、BP 神经网络、小波变换和贝叶斯等方法对问题给出求解的过程和结果。
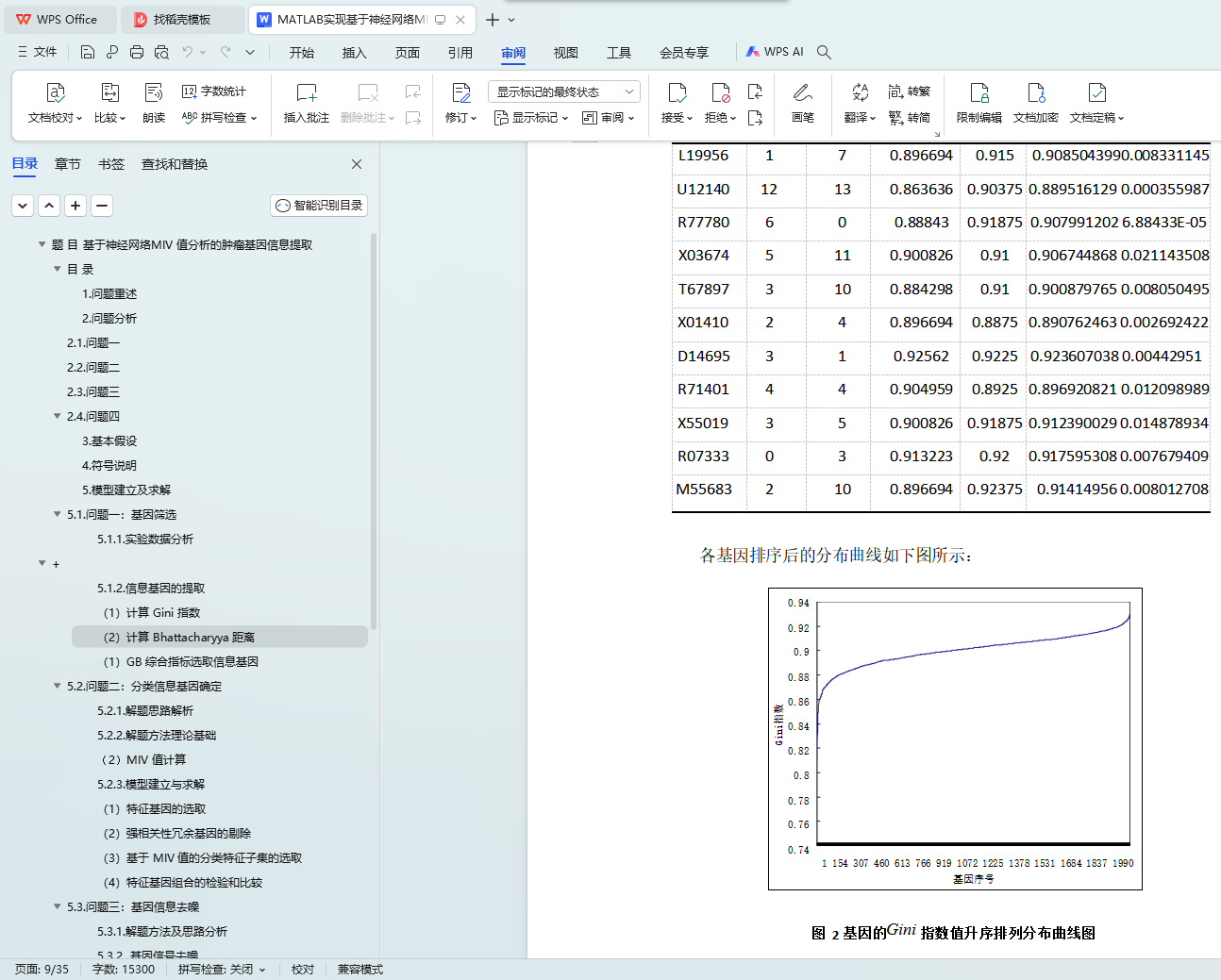
问题一采用 GB 综合指数对无关基因进行筛选。首先计算各个基因的Gini 指数和
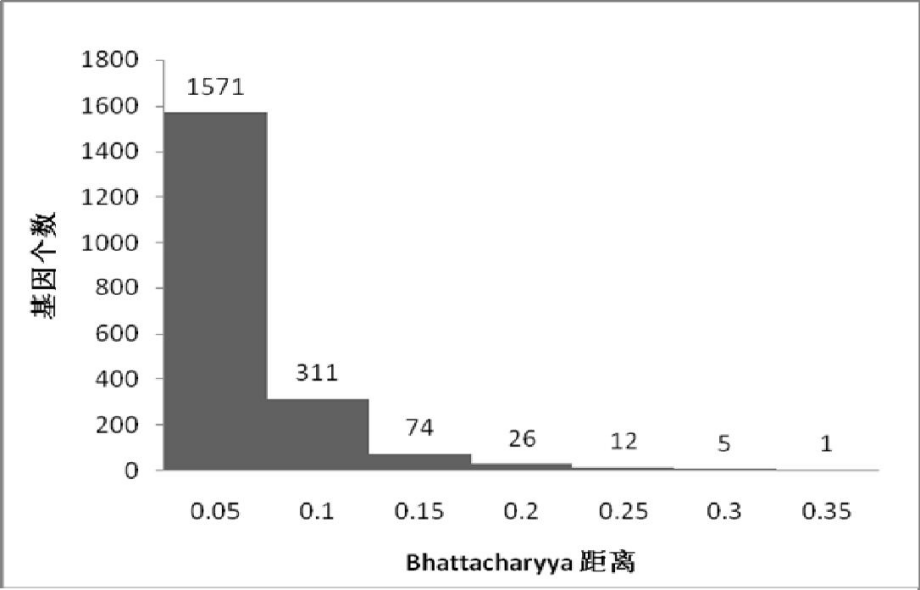
Bhattacharyya 距离,其次合理定位阈值,分别在Gini 指数排序和 Bhattacharyya 距离排序中选择 300 个优势位置的基因作为备用基因,最后选择这两组备用基因的交集作为信息基因, 共 114 个。从而降低了基因维度。
问题二结合已有文献,本着创新性和有效性的思想进行基因信息提取。
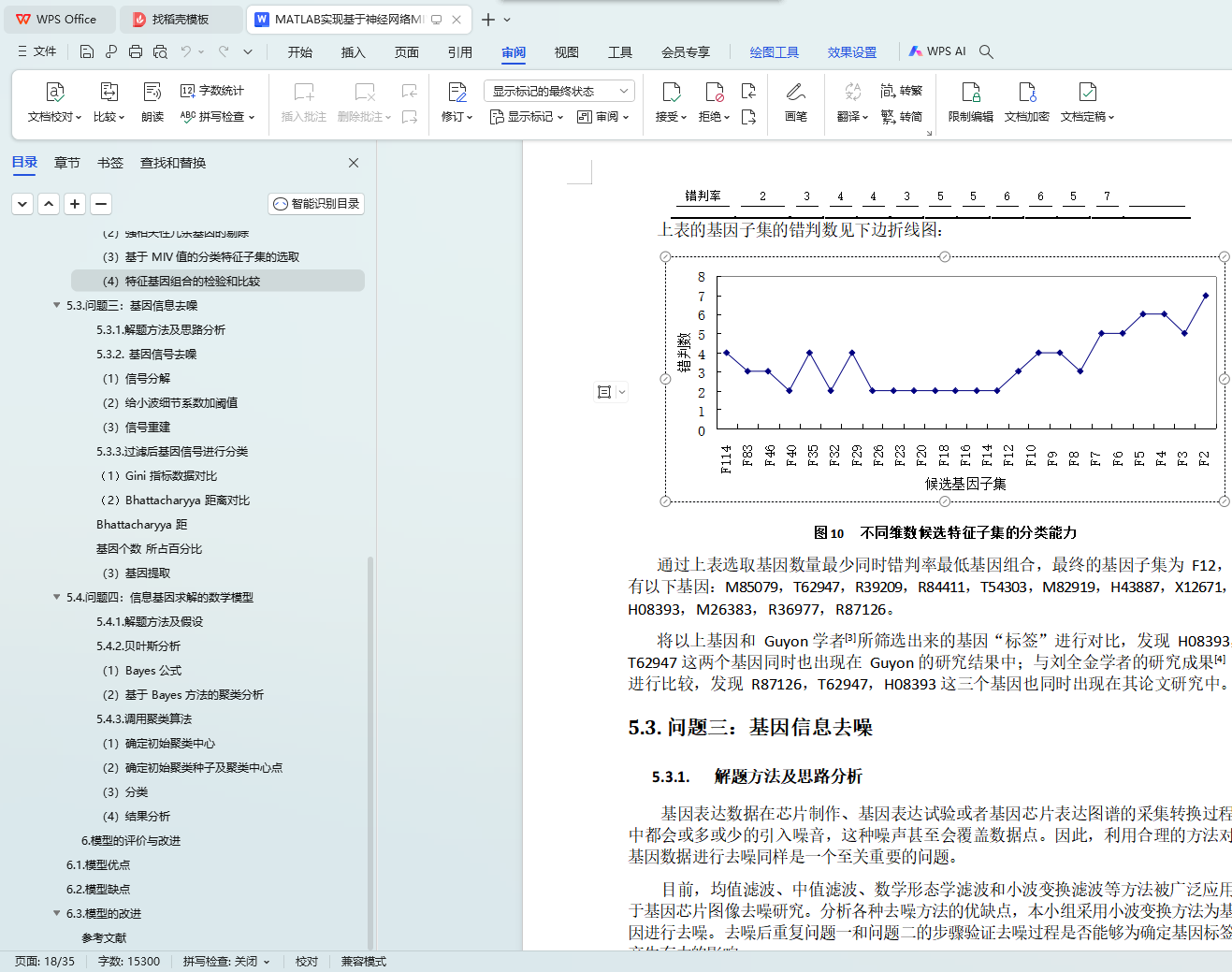
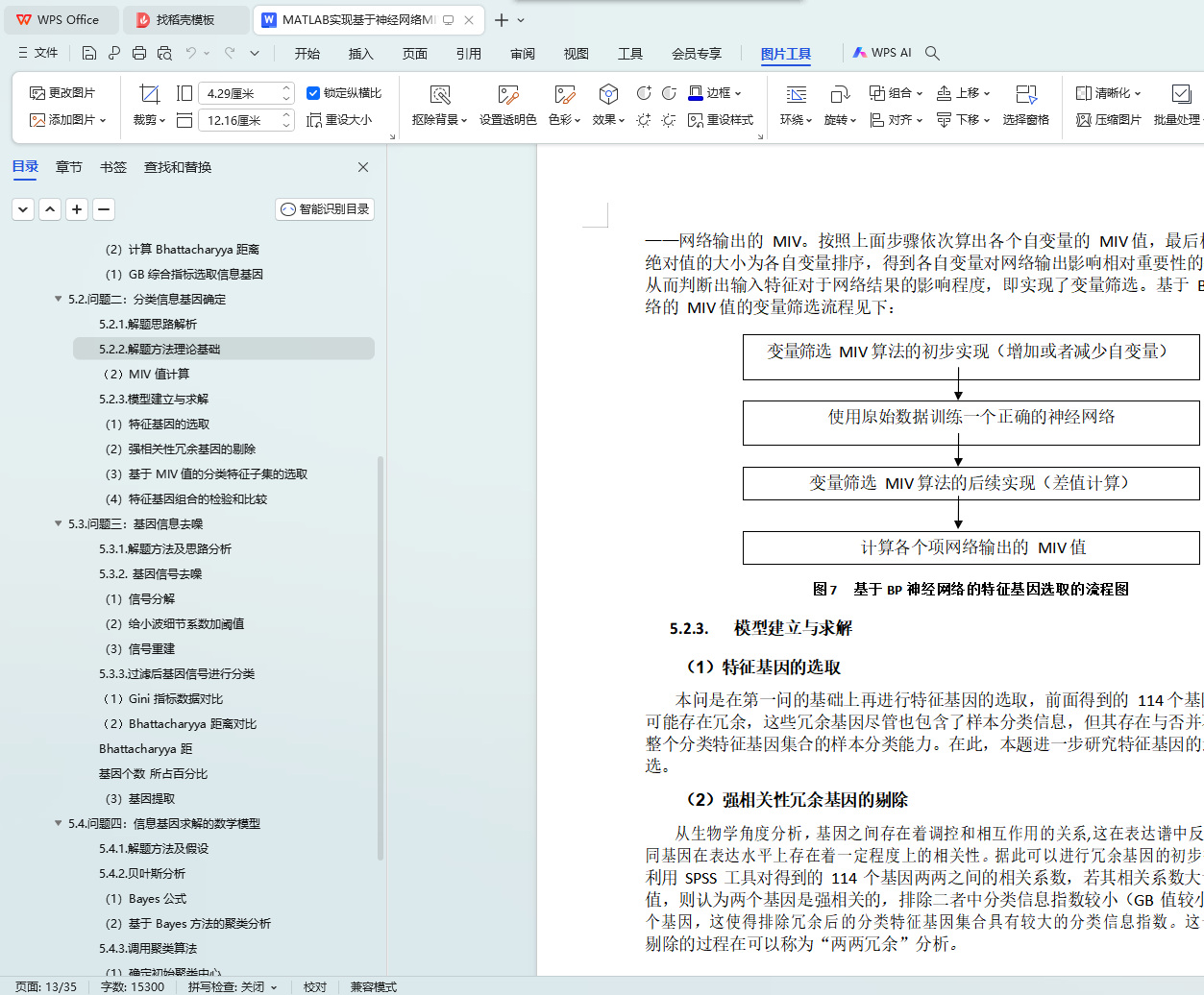
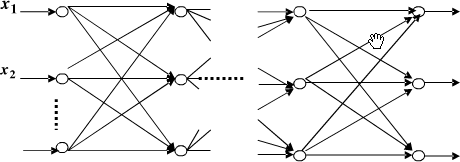
Ø 首先利用基因间的强相关性进行初步冗余基因的剔除,得到了五组特征基因组;利用 BP 神经网络对这五组基因组进行错判数计算,选取错判率最低、基因子集中基因数量最少的基因特征组作为下一步研究基因子集;
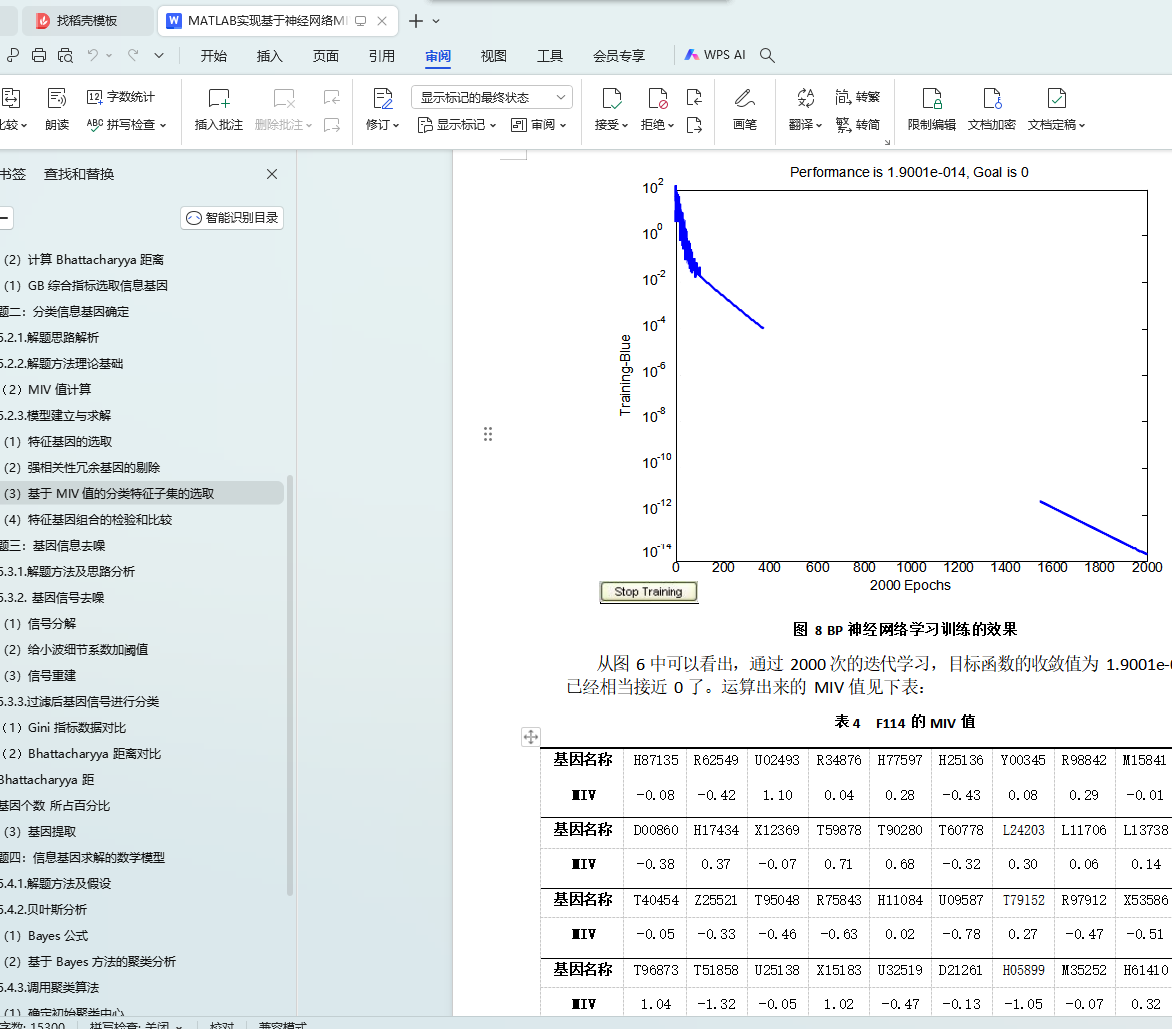
Ø 其次利用平均影响值(MIV)方法来进行筛选基因,通过计算一个基因组合中每个基因的 MIV 值,每次剔除MIV 绝对值处于后 10%的基因进行基因子集的确定;
Ø 最后利用 BP 神经网络来进行 22 个基因子集的错判数计算,最终确定含有 12 个基因的子集为最优基因组合(M85079,T62947,R39209,R84411,T54303,M82919,
H43887,X12671,H08393,M26383,R36977,R87126)。
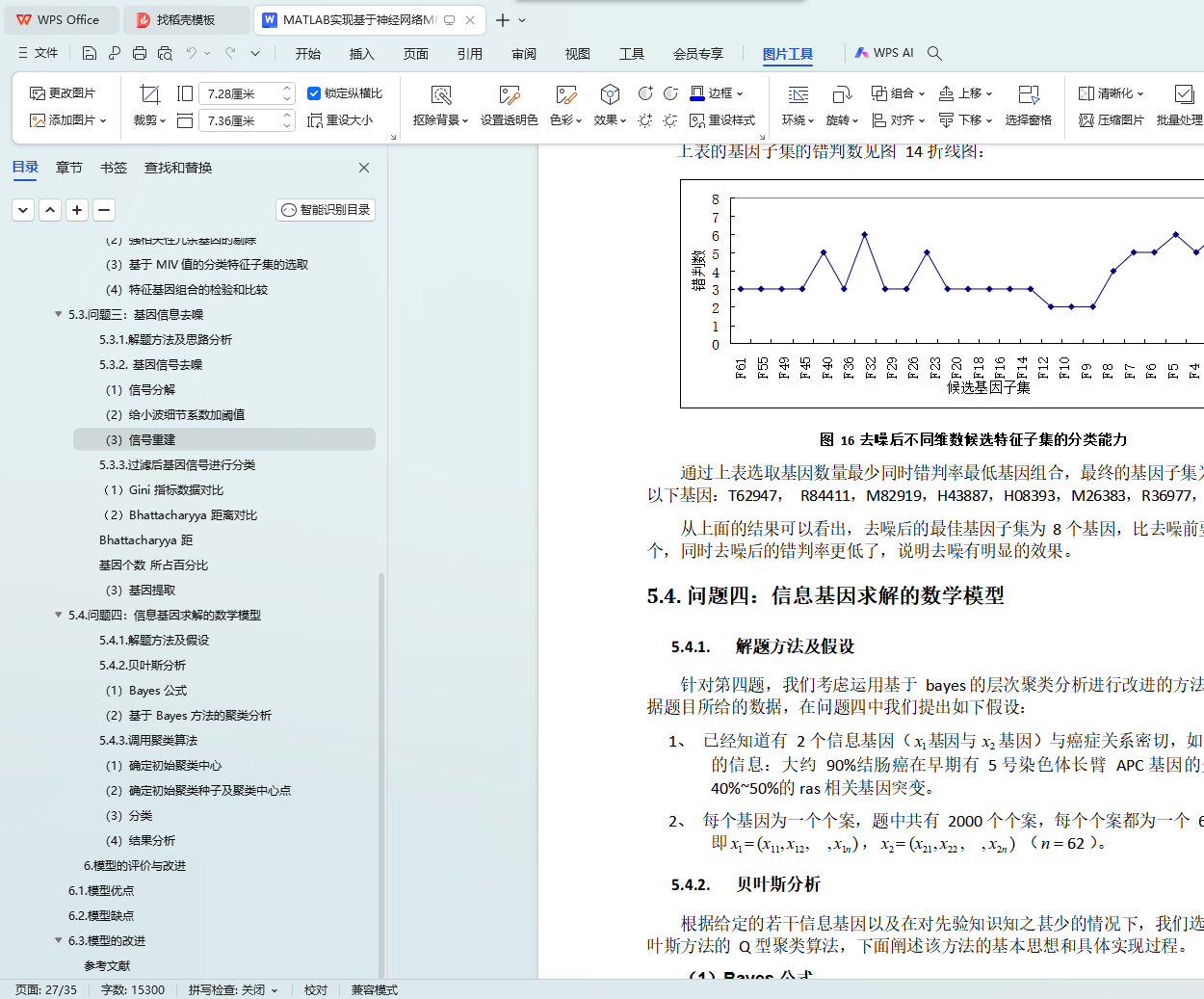
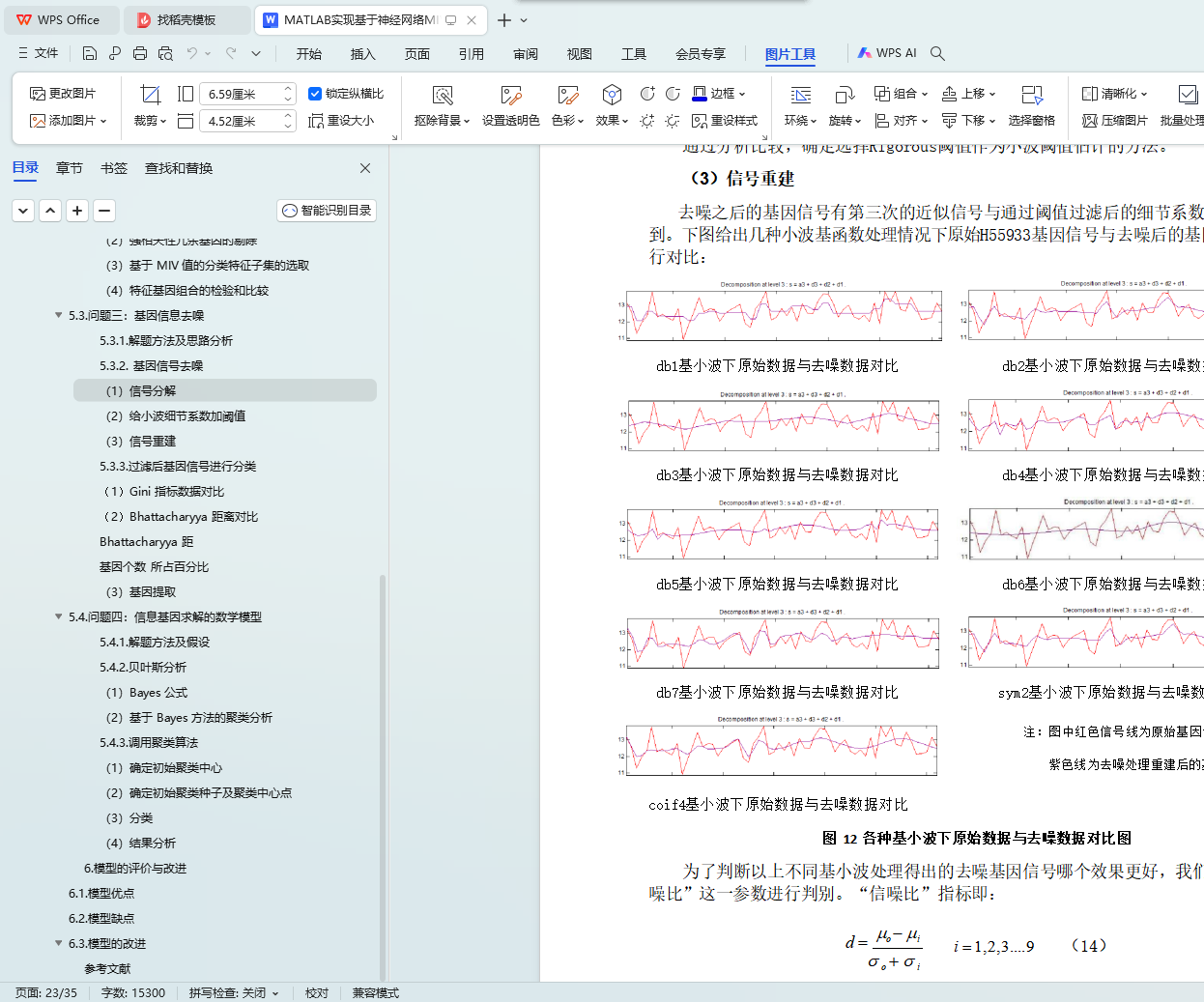
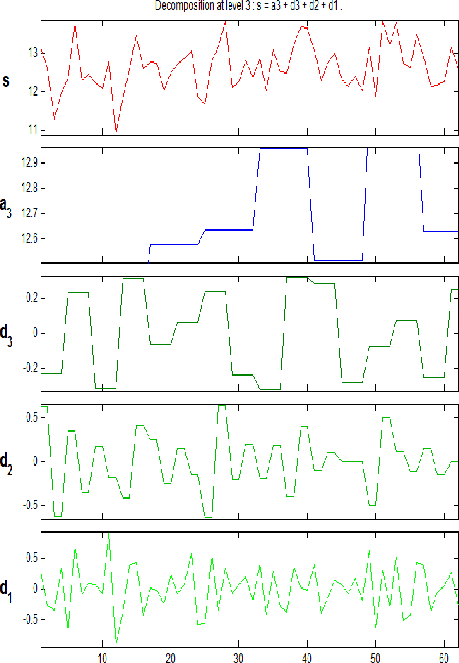
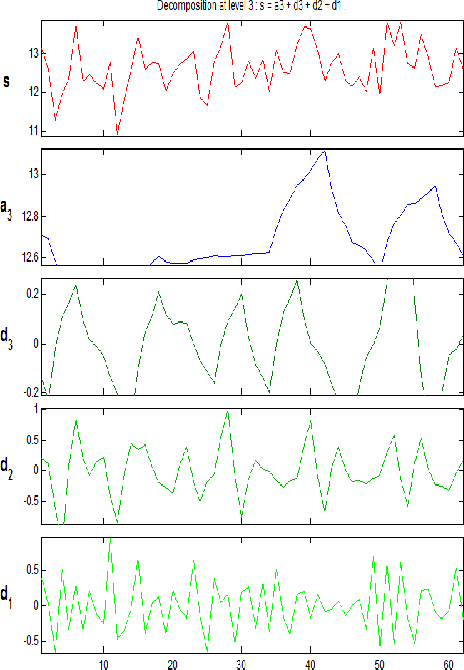
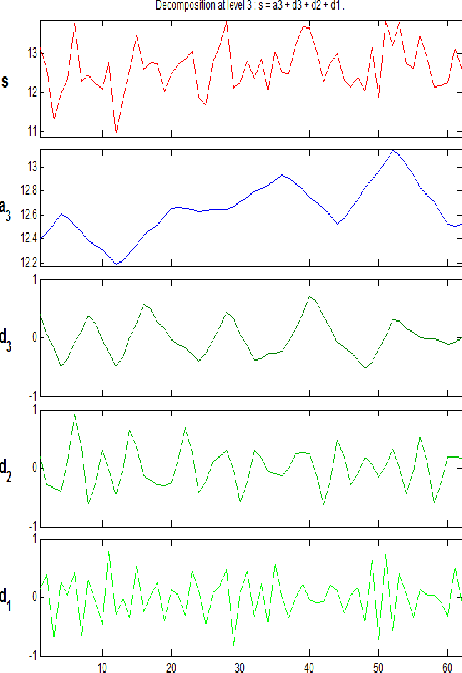
问题三将每组基因表达值看做基因信号,运用小波转换法对基因数据进行去噪,建立去噪模型。运用 MATLAB 小波工具箱对基因表达数据进行去噪处理,并运用去噪后的基因数据重新进行基因的分类、特征基因的提取。通过对比发现,去噪后的数据进行基因分类时能保留 61 个基因,比原始基因数据少 53 个,特征基因提取为 8 个。
问题四针对给定的若干信息基因,利用聚类分析原理和 Bayes 估计,通过模型建立给出探索其它未知信息基因的思想。
关键字:基因表达谱;GB 指数;MIV;BP 神经网络;小波变换;贝叶斯
目 录
1. 问题重述 1
2. 问题分析 1
2.1. 问题一 1
2.2. 问题二 1
2.3. 问题三 2
2.4. 问题四 2
3. 基本假设 2
4. 符号说明 2
5. 模型建立及求解 3
5.1. 问题一:基因筛选 3
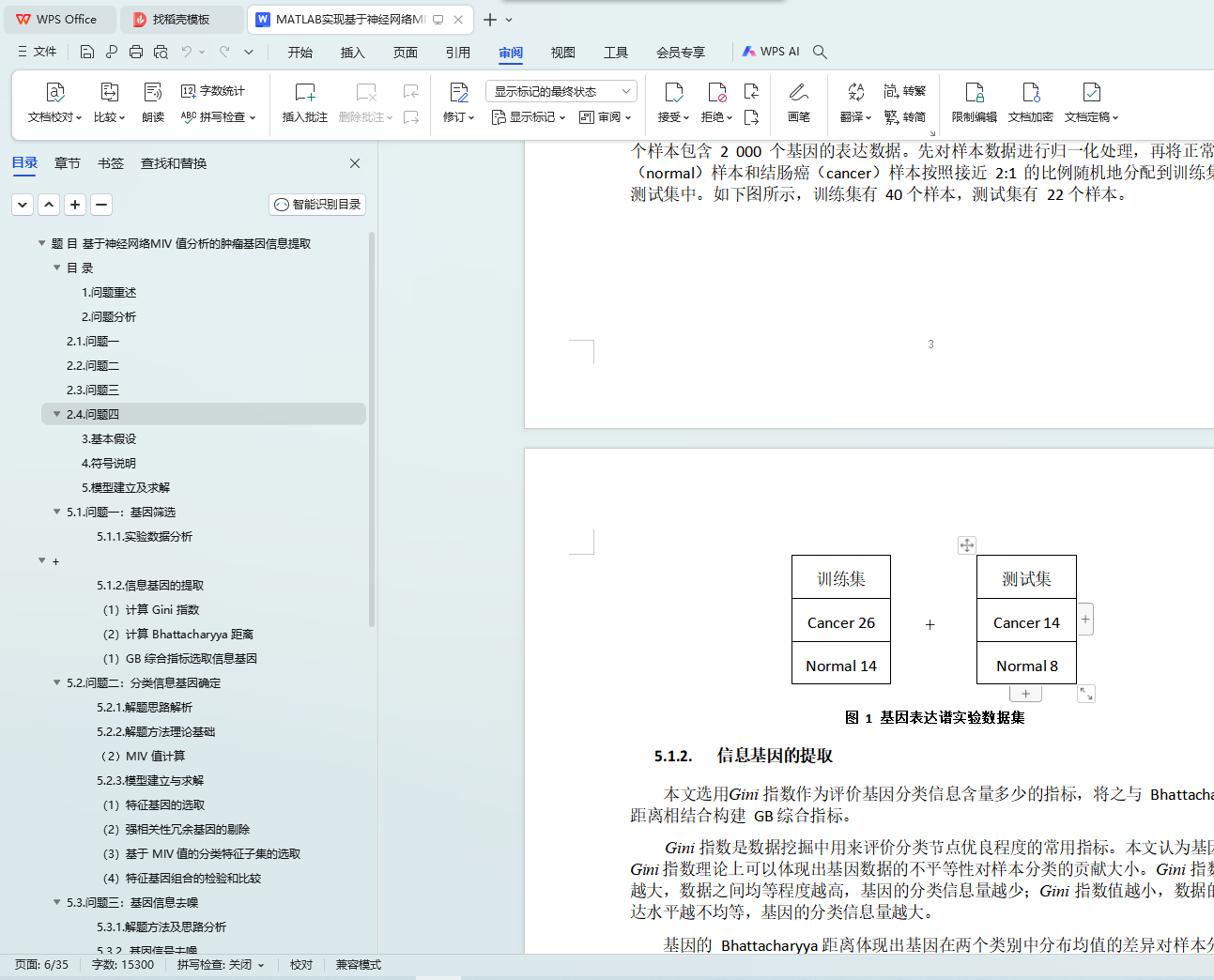
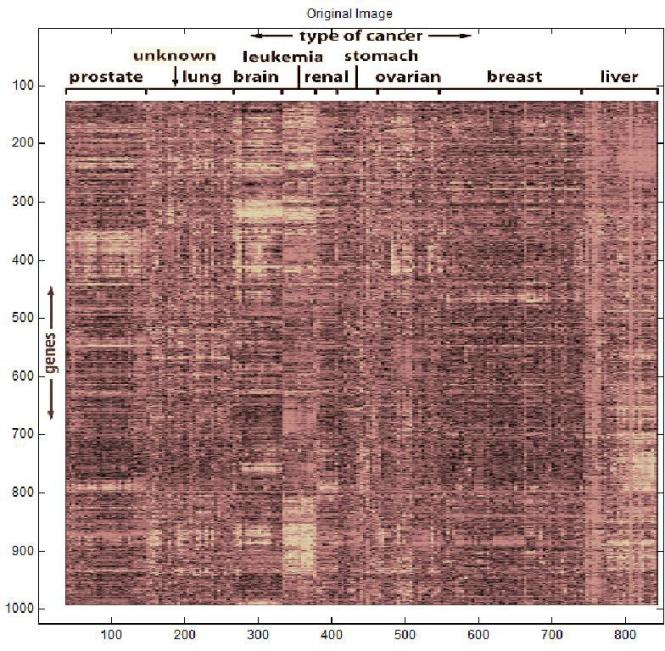
5.1.1. 实验数据分析 3
5.1.2. 信息基因的提取 4
(3)GB 综合指标选取信息基因 8
5.2. 问题二:分类信息基因确定 9
5.2.1. 解题思路解析 9
5.2.2. 解题方法理论基础 9
5.2.3. 模型建立与求解 10
5.3. 问题三:基因信息去噪 15
5.3.1. 解题方法及思路分析 15
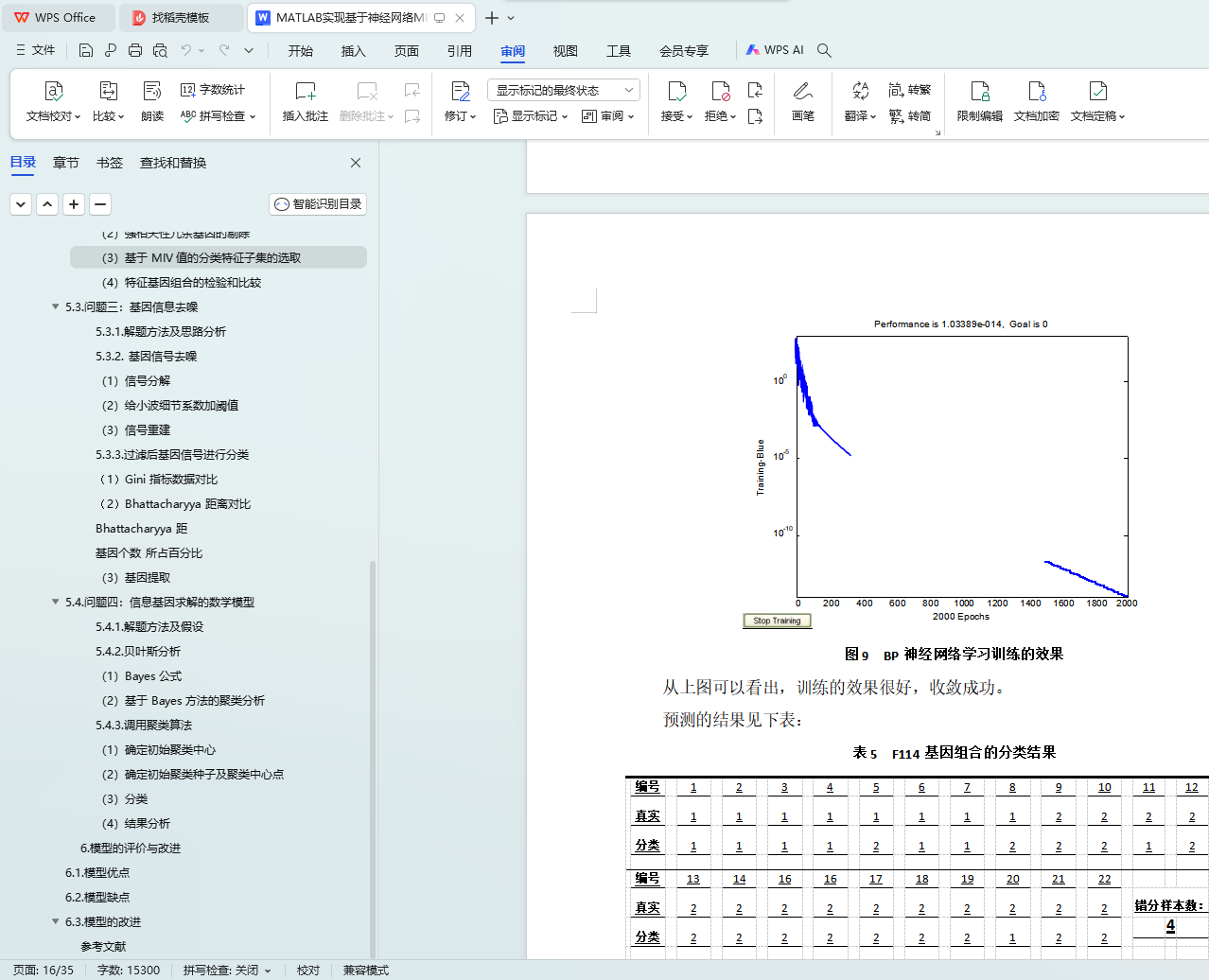
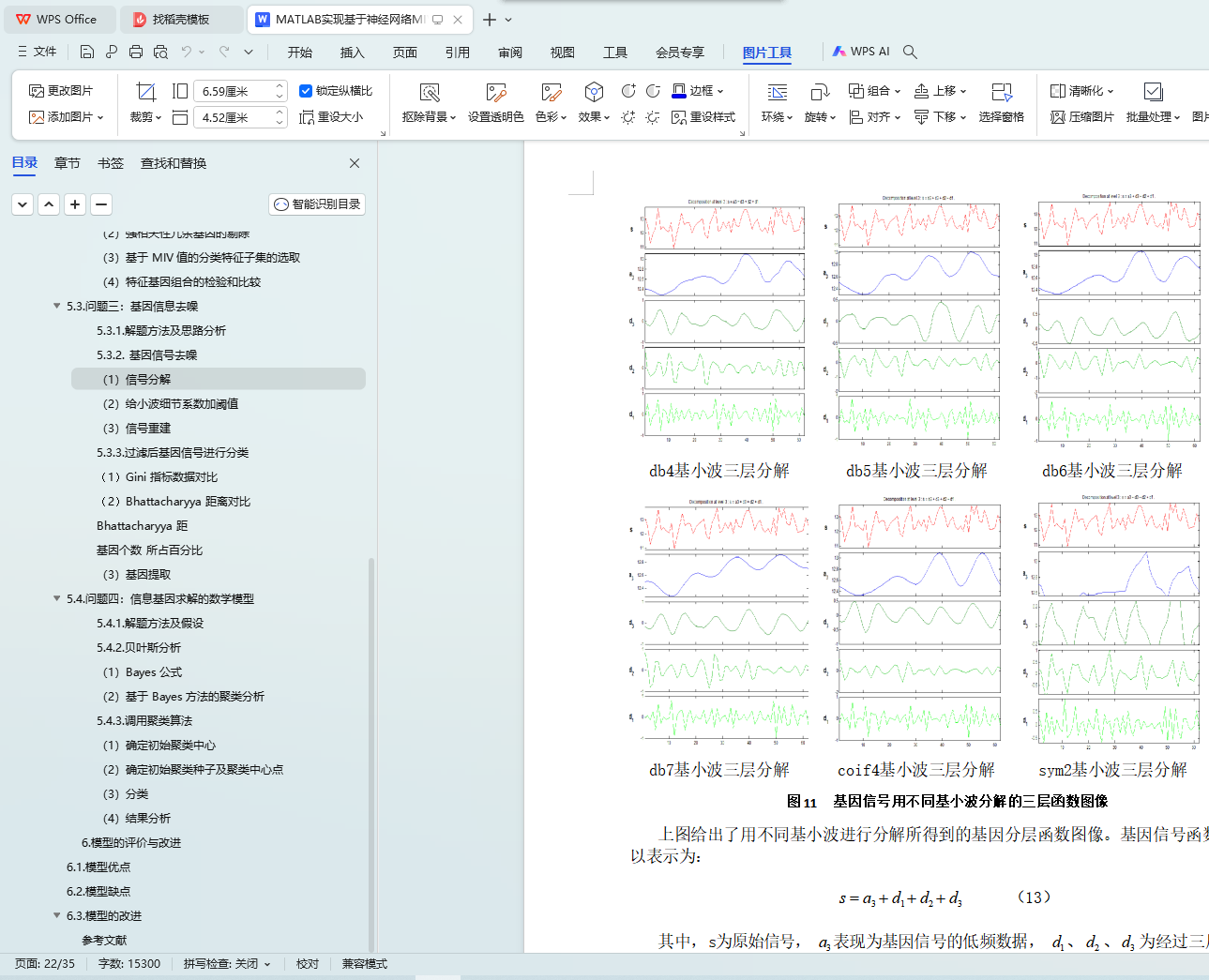
5.3.2. 基因信号去噪 16
5.3.3. 过滤后基因信号进行分类 21
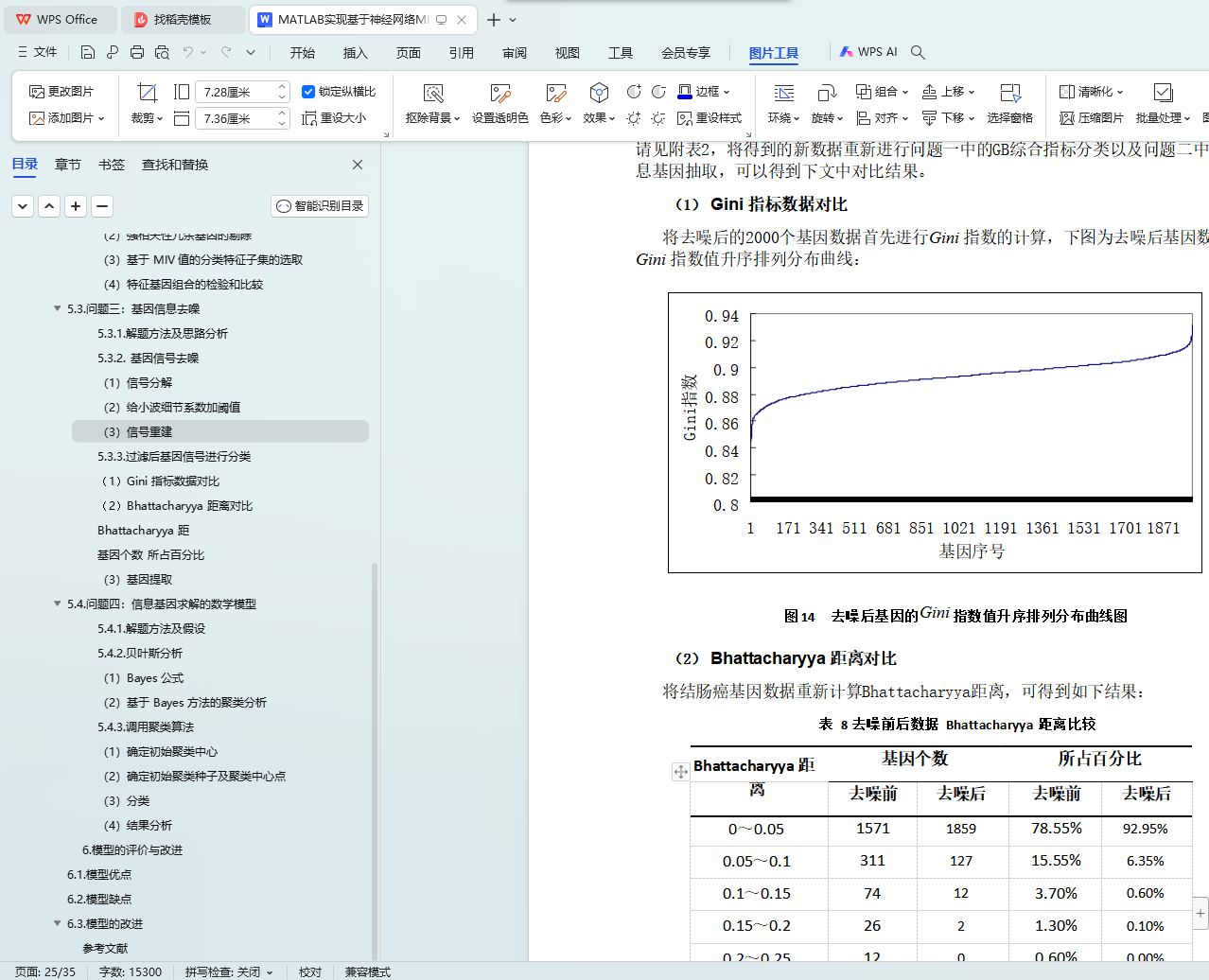
(1) Gini 指标数据对比 21
5.4. 问题四:信息基因求解的数学模型 23
5.4.1. 解题方法及假设 23
5.4.2. 贝叶斯分析 23
(2)基于 Bayes 方法的聚类算法 24
5.4.3. 调用聚类算法 25
6. 模型的评价与改进 26
6.1. 模型优点 26
6.2. 模型缺点 26
6.3. 模型的改进 27